MacroAPE 1083:HES1 f1
From FANTOM5_SSTAR
Full Name: HES1_f1
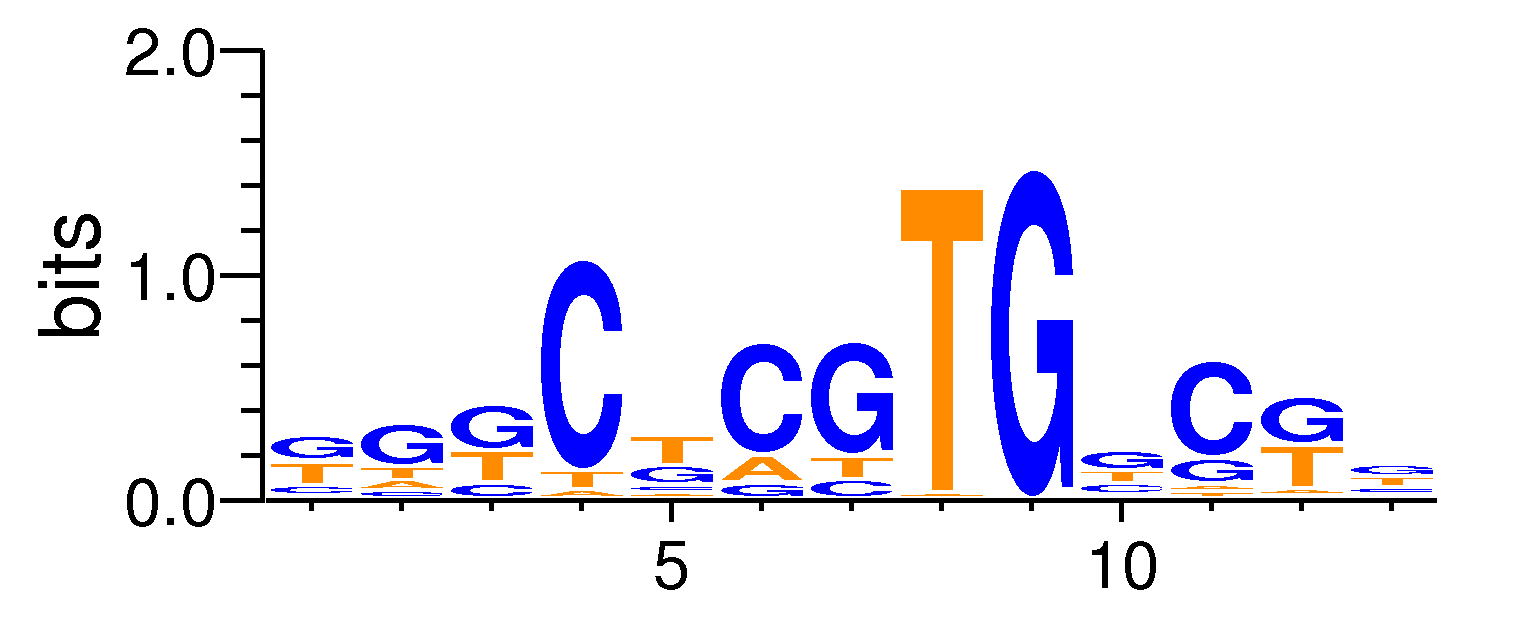
| Motif matrix | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
Target_ID | Optimal_offset | p-value | E-value | q-value | Overlap | Query consensus | Target consensus | Orientation |
|---|---|---|---|---|---|---|---|---|
| h#MA0449.1 | -1 | 5.82066e-06 | 0.00277063 | 0.00270941 | 10 | GGGCTCGTGGCGG | GGCACGTGCC | + |
| Mycn#MA0104.2 | -1 | 0.000174101 | 0.0828723 | 0.0540274 | 10 | GGGCTCGTGGCGG | CGCACGTGGC | + |
| Myc#MA0147.1 | -1 | 0.000253597 | 0.120712 | 0.0590224 | 10 | GGGCTCGTGGCGG | CGCACGTGGC | + |
| RTG3#MA0376.1 | 4 | 0.000509662 | 0.242599 | 0.0790795 | 13 | GGGCTCGTGGCGG | ATATTAGCACGTGCTTAGTC | + |
| HAC1#MA0310.1 | -3 | 0.00152914 | 0.727872 | 0.161577 | 8 | GGGCTCGTGGCGG | CACGTGTC | - |
| USF1#MA0093.1 | -3 | 0.00217272 | 1.03422 | 0.161577 | 7 | GGGCTCGTGGCGG | CACGTGG | + |
| Mycn#MA0104.2 | -3 | 0.00225443 | 1.07311 | 0.161577 | 6 | GGGCTCGTGGCGG | CACGTG | + |
| PHO4#MA0357.1 | -2 | 0.00229923 | 1.09443 | 0.161577 | 8 | GGGCTCGTGGCGG | GCACGTGC | + |
| Arnt::Ahr#MA0006.1 | -3 | 0.00242982 | 1.15659 | 0.161577 | 6 | GGGCTCGTGGCGG | TGCGTG | + |
| MYC::MAX#MA0059.1 | 0 | 0.00294575 | 1.40218 | 0.182826 | 11 | GGGCTCGTGGCGG | GAGCACGTGGT | + |
| Arnt#MA0004.1 | -3 | 0.0032765 | 1.55961 | 0.190644 | 6 | GGGCTCGTGGCGG | CACGTG | + |
| MAX#MA0058.1 | -2 | 0.00409774 | 1.95053 | 0.211936 | 10 | GGGCTCGTGGCGG | TCACGTGGTC | - |
| Gamyb#MA0034.1 | -1 | 0.00514088 | 2.44706 | 0.249302 | 10 | GGGCTCGTGGCGG | GGCGGTTGTC | - |
| TYE7#MA0409.1 | -2 | 0.00535577 | 2.54935 | 0.249302 | 7 | GGGCTCGTGGCGG | TCACGTG | - |
| ceh-22#MA0264.1 | 0 | 0.00645278 | 3.07152 | 0.25309 | 11 | GGGCTCGTGGCGG | TTTCAAGTGGT | - |
| MET32#MA0334.1 | -5 | 0.00652459 | 3.1057 | 0.25309 | 7 | GGGCTCGTGGCGG | TGTGGCG | - |
| CBF1#MA0281.1 | -2 | 0.00704413 | 3.353 | 0.262313 | 8 | GGGCTCGTGGCGG | TCACGTGC | - |
| TFAP2A#MA0003.1 | -2 | 0.00784254 | 3.73305 | 0.277748 | 9 | GGGCTCGTGGCGG | CCCCCGGGC | - |
| YGR067C#MA0425.1 | 1 | 0.00805531 | 3.83433 | 0.277748 | 13 | GGGCTCGTGGCGG | TGAAAAAGTGGGGT | - |
| MET31#MA0333.1 | -3 | 0.00876508 | 4.17218 | 0.291428 | 9 | GGGCTCGTGGCGG | GGTGTGGCG | + |
| HIF1A::ARNT#MA0259.1 | -2 | 0.0111041 | 5.28556 | 0.336579 | 8 | GGGCTCGTGGCGG | GCACGTCC | - |
| Pax4#MA0068.1 | 8 | 0.0112077 | 5.33486 | 0.336579 | 13 | GGGCTCGTGGCGG | GGGGGGGGAGTGGAGTATTGGAAATTTTTC | - |
| MBP1#MA0329.1 | -1 | 0.0122763 | 5.84354 | 0.349779 | 7 | GGGCTCGTGGCGG | TACGCGT | - |
| CREB1#MA0018.1 | -2 | 0.0146739 | 6.98476 | 0.39031 | 11 | GGGCTCGTGGCGG | CCTGGTGACGTC | + |
| CRZ1#MA0285.1 | -6 | 0.0174921 | 8.32623 | 0.449278 | 7 | GGGCTCGTGGCGG | GTGGCTTAG | - |
| INSM1#MA0155.1 | 0 | 0.017856 | 8.49945 | 0.449278 | 12 | GGGCTCGTGGCGG | TGTCAGGGGGCG | + |
| id1#MA0120.1 | 0 | 0.0196669 | 9.36146 | 0.450086 | 12 | GGGCTCGTGGCGG | TTTTCCTTTTCG | + |
| RREB1#MA0073.1 | 3 | 0.0202924 | 9.65919 | 0.450086 | 13 | GGGCTCGTGGCGG | TGGGGGGGGGTGGTTTGGGG | - |
| RSC3#MA0374.1 | -3 | 0.0208945 | 9.94577 | 0.450086 | 7 | GGGCTCGTGGCGG | CGCGCGG | + |
| YPR022C#MA0436.1 | -5 | 0.0208945 | 9.94577 | 0.450086 | 7 | GGGCTCGTGGCGG | CGTGGGG | - |