Full Name: HES1_f1
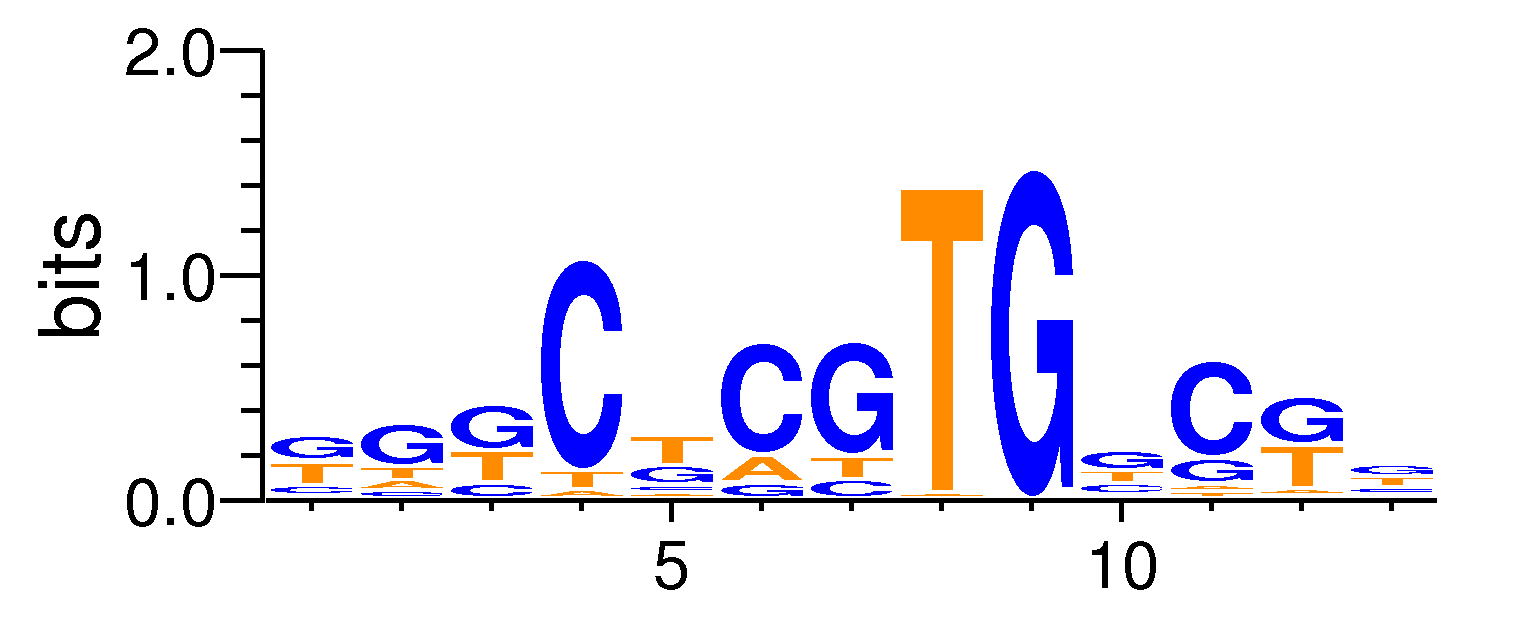
| Motif matrix
|
| PO | A | C | G | T | | 1 | 1.5166666666666677 | 4.433333333333324 | 11.666666666666677 | 10.383333333333324 | | 2 | 3.9666666666666757 | 2.9166666666666763 | 15.983333333333324 | 5.133333333333324 | | 3 | 0.0 | 4.783333333333324 | 13.65 | 9.566666666666677 | | 4 | 1.05 | 24.733333333333324 | 0.0 | 2.2166666666666677 | | 5 | 2.5666666666666678 | 3.266666666666676 | 8.75 | 13.416666666666677 | | 6 | 4.8999999999999995 | 20.183333333333323 | 2.9166666666666763 | 0.0 | | 7 | 0.0 | 3.5 | 20.416666666666675 | 4.083333333333324 | | 8 | 0.5833333333333324 | 0.0 | 0.0 | 27.416666666666675 | | 9 | 0.0 | 0.0 | 28.0 | 0.0 | | 10 | 1.9833333333333325 | 6.6499999999999995 | 11.9 | 7.466666666666676 | | 11 | 1.5166666666666677 | 19.599999999999998 | 5.366666666666676 | 1.5166666666666677 | | 12 | 1.9833333333333325 | 0.9333333333333323 | 13.299999999999999 | 11.783333333333324 | | 13 | 3.0916666666666757 | 6.125 | 9.975 | 8.808333333333323 |
|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
No results for this motif
