MacroAPE 1083:DMC1 Testis-DMC1-ChIP-Seq/Homer
From FANTOM5_SSTAR
Full Name: DMC1_Testis-DMC1-ChIP-Seq/Homer
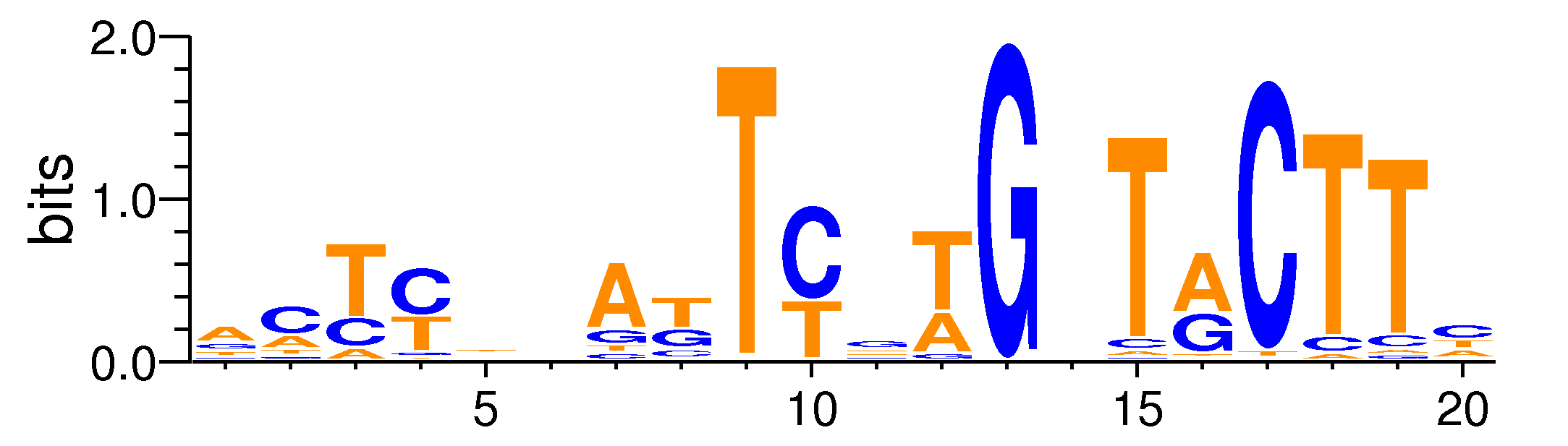
| Motif matrix | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
Target_ID | Optimal_offset | p-value | E-value | q-value | Overlap | Query consensus | Target consensus | Orientation |
|---|---|---|---|---|---|---|---|---|
| Pax4#MA0068.1 | 8 | 0.0040164 | 1.91181 | 1 | 20 | ACTCTAATTCGTGCTACTTC | GGGGGGGGAGTGGAGTATTGGAAATTTTTC | - |
| RTG3#MA0376.1 | 0 | 0.00940562 | 4.47708 | 1 | 20 | ACTCTAATTCGTGCTACTTC | GACTAAGCACGTGCTAATAT | - |
| SMP1#MA0383.1 | 4 | 0.0143615 | 6.83609 | 1 | 17 | ACTCTAATTCGTGCTACTTC | TGCTAATTTAATTATAGGTAA | - |
| NKX3-1#MA0124.1 | -13 | 0.0165397 | 7.87289 | 1 | 7 | ACTCTAATTCGTGCTACTTC | ATACTTA | + |